「タンパク質を究める」
この世界に存在する物質の中で、「タンパク質」ほど素晴らしい物質は存在しないのではないでしょうか?
タンパク質は、単なる物質なのに、まるで生きているかのように動き、様々な物質に結合したり、様々な物質を作り出したりすることができます。生物の中には、このようなタンパク質が無数に存在し、ダイナミックに動いています。私たちが動いたり、考えたりすることができるのは、タンパク質のおかげです。生命体の生き生きとした動きの原動力は、単なる物質であるタンパク質の生きているかのような動きなのです。
それでは、単なる物質であるタンパク質は、一体どのようにして、生きているかのように動くことができるのでしょうか? こうした疑問に答えるためには、タンパク質の基本的な性質を、物理学的な観点から理解することが必要です。このような研究が、タンパク質の物性研究です。
研究の方法
タンパク質の物理的な性質を理解するためには、手段を選ばず、ありとあらゆる手法を駆使して研究を行います。生化学、分子生物学、物理化学、熱力学、統計力学、バイオインフォマティクスなどを総動員します。
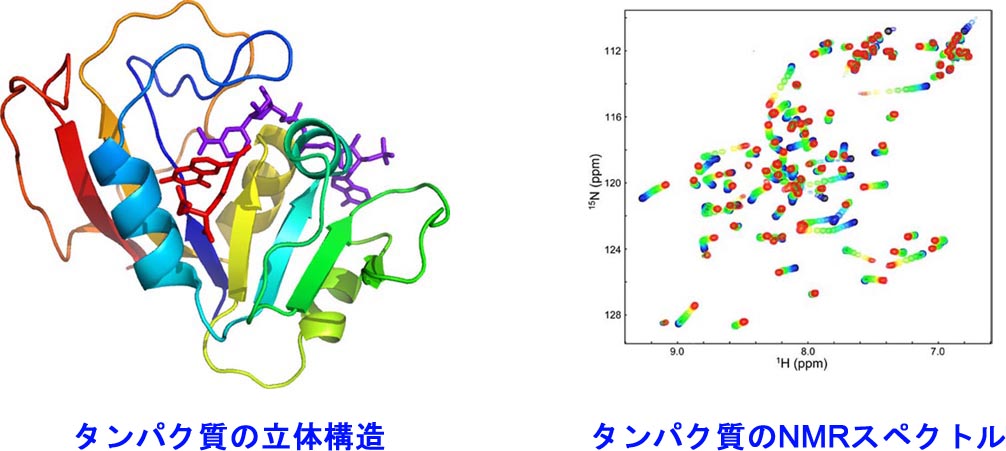
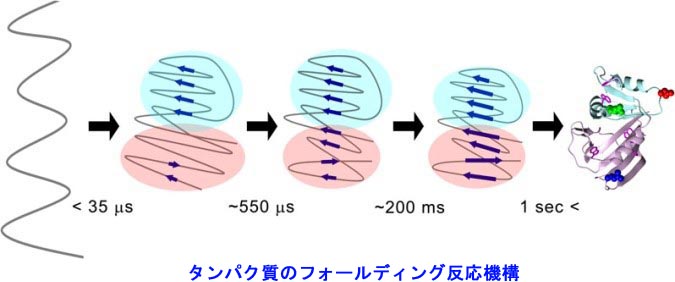
まず、遺伝子組み換え技術やタンパク質工学技術によって研究対象のタンパク質を作成し、高純度精製します。次に、得られたタンパク質試料を徹底的に研究します。例えば、タンパク質の立体構造、変性、フォールディング、安定性、ダイナミクス、標的分子との結合反応、酵素反応などを詳細に調べます。用いる実験手法は、核磁気共鳴(NMR)法、X線結晶構造解析、X線溶液散乱法、速度論・緩和法(ストップトフロー法)、分光(蛍光)測定、熱測定などです。また、分子動力学シミュレーションなどの理論的研究も行います。
実験と理論から得られたデータを詳細に解析し、タンパク質構造の時間的・空間的変化を、状態、エネルギー、相互作用といった観点から記述し、メカニズムを解明します。可能ならば、ピコ秒(10-12 秒)までの時間分解能と、0.1 nm(10-10 m)までの空間分解能でタンパク質の動きを解明することに挑戦します。以上のような研究を通して、タンパク質を究めることを目指しています。
よく研究されているタンパク質としては、形状で分類すると、球状タンパク質や天然変性タンパク質などがあります。また、機能で分類すると、結合タンパク質、酵素、モータータンパク質などがあります。
波及効果、社会的貢献
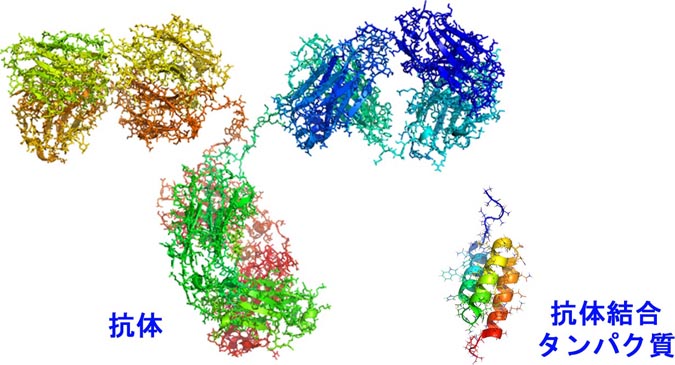
タンパク質は、生命体の中のあらゆる場所で働いているので、病気にも密接に関係しています。タンパク質の構造や物性を明らかにすることは、病気の発症機構を解明するだけでなく、病気を治す方法を開発する上でも非常に重要です。例えば、狂牛病、アルツハイマー病、パーキンソン病などの神経変性疾患や、食物アレルギーなどは、タンパク質の物性と密接に関係しています。また、最近では、抗体などのタンパク質を薬として用いたタンパク質医薬品が次々と実用化されており、次世代型医薬品として期待されています。
タンパク質は様々な物質を作ったり分解したりすることもできるので、食品加工や物質生産など、様々な産業にも利用されています。特に、現在期待されているのがバイオエネルギー生産です。軽油に相当するアルカンを合成するタンパク質(酵素)を使って、シアノバクテリアなどの植物内で大量のアルカンを作り出すことができれば、エネルギー問題の解決の一助になるかもしれません。しかも、このような植物は、燃料を燃やして発生した二酸化炭素を使って再びアルカンを作ることができるので、二酸化炭素量の増加も防げます。
さらに、タンパク質を設計する技術も進展しており、21世紀のうちに、医療や産業に役立つタンパク質を計算機上で自由自在に設計し、実際に使えるような時代が来ると期待されています。もしそれが可能になったら、私たちの生活は一変するでしょう。このようなタンパク質の設計技術は、生命科学の時代と呼ばれる21世紀の最大の偉業の一つとして語り継がれるかもしれません。
参考文献
Yang, K., Arai, M., & Wright, P.E. (2020) Determining binding kinetics of intrinsically disordered proteins by NMR spectroscopy. Methods Mol. Biol. in press.
Kunihara, T., Hayashi, Y., & Arai, M. (2019) Conformational diversity in the intrinsically disordered HIV-1 Tat protein induced by zinc and pH. Biochem. Biophys. Res. Commun. 509(2), 564-569.
Kenri, T., Kawakita, Y., Kudo, H., Matsumoto, U., Mori, S., Furukawa, Y., Tahara, Y.O., Shibayama, K., Hayashi, Y., Arai, M., & Miyata, M. (2019) Production and characterization of recombinant P1 adhesin essential for adhesion, gliding, and antigenic variation in the human pathogenic bacterium, Mycoplasma pneumoniae. Biochem. Biophys. Res. Commun. 508(4), 1050-1055.
Kujirai, J., Nanba, S., Kadowaki, T., Oka, Y., Nishiyama, Y., Hayashi, Y., Arai, M., & Hihara, Y. (2018) Interaction of the GntR-family transcription factor Sll1961 with thioredoxin in the cyanobacterium Synechocystis sp. PCC 6803. Scientific Reports 8, 6666.
Takenaka, T., Nakamura, T., Yanaka, S., Yagi-Utsumi, M., Chandak, M.S., Takahashi, K., Paul, S., Makabe, K., Arai, M., Kato, K., & Kuwajima, K. (2017) Formation of the chaperonin complex studied by 2D NMR spectroscopy. PLoS ONE 12(10), e0187022.
Otosu, T., Ishii, K., Oikawa, H., Arai, M., Takahashi, S., & Tahara, T. (2017) Highly heterogeneous nature of the native and unfolded states of B domain of protein A revealed by two-dimensional fluorescence lifetime correlation spectroscopy. J. Phys. Chem. B 121(22), 5463-5473.
Haberz, P., Arai, M., Martinez-Yamout, M.A., Dyson, H.J., & Wright, P.E. (2016) Mapping the interactions of adenoviral E1A proteins with the p160 nuclear receptor coactivator binding domain of CBP. Protein Sci. 25(12), 2256-2267.
Arai, M., Sugase, K., Dyson, H.J., & Wright, P.E. (2015) Conformational propensities of intrinsically disordered proteins influence the mechanism of binding and folding. Proc. Natl. Acad. Sci. USA 112(31), 9614-9619.
Oikawa, H., Kamagata, K., Arai, M., & Takahashi, S. (2015) Complexity of the folding transition of the B domain of protein A revealed by the high-speed tracking of single-molecule fluorescence time series. J. Phys. Chem. B 119(20), 6081-6091.
Ohori, Y., Okazaki, H., Watanabe, S., Tochio, N., Arai, M., Kigawa, T., & Nishimura, C. (2014) Flexible and rigid structures in HIV-1 p17 matrix protein monitored by relaxation and amide proton exchange with NMR. Biochim. Biophys. Acta 1844(3), 520-526.
Oikawa, H., Suzuki, Y., Saito, M., Kamagata, K., Arai, M., & Takahashi, S. (2013) Microsecond dynamics of an unfolded protein by a line confocal tracking of single molecule fluorescence. Scientific Reports 3, 2151.
新井宗仁(2013)「タンパク質の揺らぎと機能 〜結合と触媒〜」『揺らぎ・ダイナミクスと生体機能 〜物理化学的視点から見た生体分子〜』(寺嶋正秀 編)第17章 pp.267-280, 化学同人(ISBN-10: 9784759815108)Arai, M., Ferreon, J.C., & Wright, P.E. (2012) Quantitative analysis of multisite protein ligand interactions by NMR: binding of intrinsically disordered p53 transactivation subdomains with the TAZ2 domain of CBP. J. Am. Chem. Soc. 134(8), 3792-3803.
Arai, M., Iwakura, M., Matthews, C.R., & Bilsel, O. (2011) Microsecond subdomain folding in dihydrofolate reductase. J. Mol. Biol. 410(2), 329-342.
Arai, M., Dyson, H.J., & Wright, P.E. (2010) Leu628 of the KIX domain of CBP is a key residue for the interaction with the MLL transactivation domain. FEBS Lett. 584(22), 4500-4504.
Lee, C.W., Ferreon, J.C., Ferreon, A.C.M, Arai, M., & Wright, P.E. (2010) Graded enhancement of p53 binding to CREB-binding protein (CBP) by multisite phosphorylation. Proc. Natl. Acad. Sci. USA 107(45), 19290-19295.
Arai, M., & Kuwajima, K. (2000) Role of the molten globule state in protein folding. Adv. Protein Chem. 53: 209-282.